



如果您无法下载资料,请参考说明:
1、部分资料下载需要金币,请确保您的账户上有足够的金币
2、已购买过的文档,再次下载不重复扣费
3、资料包下载后请先用软件解压,在使用对应软件打开
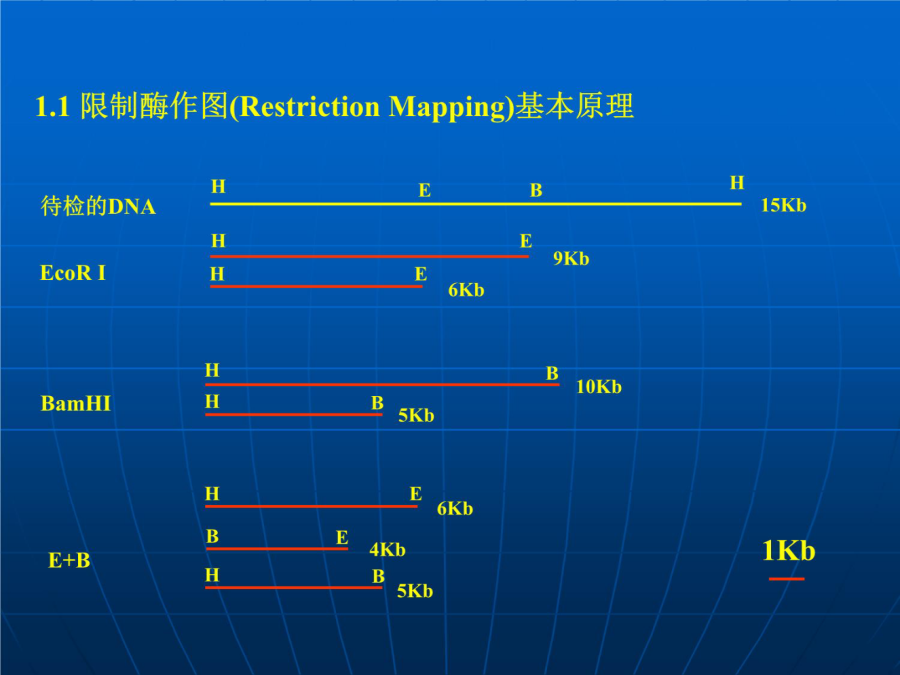
第十章物理作图(physicalmapping)物理作图的方法1.限制性作图 它是将限制性酶切位点标定在DNA分子的相对位置. 其只能应用于较小的DNA分子,其上限取决于作图分子中限制酶位点的频率. 通常采用的6碱基对识别顺序的限制酶仅适合50Kb以下DNA分子的精确作图.方法:缺点 通常,部分限制酶消化为构建完整的图谱提供了必需的信息。如果有多个限制位点集中、这种分析方法就显得笨拙,因为要考虑的不同片段太多。 改进方法: 在部分消化前将放射性或其他类型的标记物加到要分析的DNA分子两端,结果很多部分限制酶消化产物成为不可见的,因为它们不含有末端片段,因此在琼脂糖凝胶上对标记物进行筛选时不会显现。我们可以利用“可见的”部分限制片段的大小,确定出那些未定位的切点与DNA分子末端的相对位置。限制因素1.2限制酶作图的改进 脉冲凝胶电泳(pulsed-fieldgelelectrophoresis) 稀有切点限制图绘制 具有7个或8个核等酸识别序列的酶4096bp,16384BP,65536bp 识别序列包含靶DNA中稀少基序的酶基因组明显缺少某些基序:例如,人类基因组中5‘GC3’序列很稀少,这是因为人类细胞中含有一种酶,能够向这一序列中的胞嘧啶核苷的5位碳原于添加甲基基团,产生的5甲基胞嘧啶不稳定.容易脱氨基产生胸腺嘧啶。 SmaI5‘CCCGGG78kb BssHII:5’GCGCGC3:390kb频 NotI:5‘GCCCGGCCGC3’平均约每10Mb才有一个位点。 稀有切点限制图绘制注意事项: 识别顺序越长产生的片段越大; 识别位点碱基的组成影响限制性片段的大小; 如人类基因组A/T比例接近60/%,选用高比例A/T位点限制酶,产生的片段多于高比例G/C识别位点酶 有些限制酶识别位点较长 如酵母的Ⅰ-Sce识别顺序为18bp, 如果高等真核生物基因组中无此序列,可通过转座子转座和噬菌体转导的方法将其引入代测基因组中. 基因组DNA的甲基化状态 如人类活细胞基因组中大量5’-CG-3’顺序的胞嘧啶被甲基化,使许多含有5’-CG-3’顺序的内切酶很少找到可切位点. 2.1大片段DNA的克隆载体 YAC:酵母人工染色体230-1700Kb BAC:细菌人工染色体300Kb PAC:P1人工染色体300Kb Fosmids:30Kb2.2重叠群组建 重叠群:相互重叠的DNA片段组成的物理图. 重叠群的组建方法 染色体步移法指纹作图法 指纹:系指确定DNA样品所具有的DNA片段; 一个克隆的指纹表示了该克隆所具有的限定的顺序特征. 常用的指纹分析方法: A限制性带型(restrictionpatterns)指纹 B重复顺序DNA指纹(repetitiveDNAfingerprints) CSTS目录作图(STScontentmapping) D重复DNAPCR(repetitiveDNAPCR)或分散重复顺序PCR(interspersedrepeatelementPCR,IRE-PCR)指纹 3.荧光原位杂交 (Fish,Fluorescentinsituhybridization)染色体上的杂交位点提供DNA探针序列的定位信息。应用该方法时,须打开维持染色体DNA螺旋结构。只有这样染色体DNA才能与探针杂交。变性染色体DNA而不破坏其形态的标准方法是将染色体干燥在玻璃载玻片上,再用甲酰胺处理。 因为放射性标记很难同时满足灵敏度和分辨率这两个原位杂文成功的必要条件。因此,相隔1Mb才能作为分开的杂文信号被分辨出来 机械伸展的染色体通过改变从中期细胞核中分离染色体的方法而获得离心产生的离心力可将将染色体伸展到正常长度的20倍。每条染色体仍能识别。这样,相隔200-300KB的标记 非中期染色体:间期 4.序标位作图 (STS,SequenceTagedSite)4.1STS作图原理条件4.2寻找STS的方法: 表达顺序标签(expressedsequencetag,EST) 从cDNA中找到的小段顺序,但基因家族成员间共有的序列不能用于STS。 SSLP(simplesequencelengthpolymorphisrns,SSLPs) 随机基因组顺序EST 1990年,Brenneer等首先提出人类基因组大规模cDNA测序计划以来,Adams等最先采纳并最早建立了表达序列标记技术,他们随机挑选cDNA克隆进行单向、一步大规模测序,将所获得的部分cDNA序列称为EST。 EST是一个cDNA克隆快速大规模测序后所获得的3’端和5’端部分cDNA随机片段,每个EST长度约200~600bp,代表了一个单拷贝基因的部分cDNA表达序列。 由于大多数EST的长度不足400bp,说明一个基因转录本的c

王子****青蛙
 实名认证
实名认证
 内容提供者
内容提供者
最近下载
最新上传
浙江省宁波市2024-2025学年高三下学期4月高考模拟考试语文试题及参考答案.docx
汤成难《漂浮于万有引力中的房屋》阅读答案.docx
四川省达州市普通高中2025届第二次诊断性检测语文试卷及参考答案.docx
山西省吕梁市2025年高三下学期第二次模拟考试语文试题及参考答案.docx
山西省部分学校2024-2025学年高二下学期3月月考语文试题及参考答案.docx
山西省2025年届高考考前适应性测试(冲刺卷)语文试卷及参考答案.docx
全国各地市语文中考真题名著阅读分类汇编.docx
七年级历史下册易混易错84条.docx
湖北省2024-2025学年高一下学期4月期中联考语文试题及参考答案.docx
黑龙江省大庆市2025届高三第三次教学质量检测语文试卷及参考答案.docx


















